

大多数局部前列腺癌是可以治愈,但转移性疾病患者的5年生存率低至30%。转移性前列腺癌是临床的重要挑战,主要是因为其病变高度异质且难以活检。
目前,传统药物治疗无法抑制肿瘤进展,最终会产生耐药性。因此迫切需要提高我们对转移性前列腺癌的发现和治疗。
近日,来自加拿大多伦多大学癌症中心的研究团队通过ctDNA免疫沉淀结合新一代测序 (cfMeDIP-seq),分别分析了来自局部和转移性前列腺癌患者的60和175个血浆DNA甲基化组,捕获反映异质疾病生物学的变化。
结果表明:ctDNA甲基化组可以观察到转移样本中的整体高甲基化,以及着丝粒周围区域的低甲基化,所以可以以98.9%的预测准确度区分不同的疾病类型。
ctDNA甲基化可以高精度地区分不同的疾病状态,突出其作为疾病监测和预后的微创策略的潜力。
原文发表在《Nature communications》上,题目为“The cell-free DNA methylome captures distinctions between localized and metastatic prostate tumors”。
首先,了解前列腺癌进展过程中cfDNA甲基化谱的变化。
研究团队共收集了包括分别来自原发性和转移性前列腺癌患者的30个和103 个血浆样本,通过cfMeDIP数据捕获局部与转移性前列腺癌患者血浆样本中的ctDNA甲基化和片段化变化。
来自所有血浆样品的甲基化DNA片段被沉淀并进行配对末端DNA测序,发现ctDNA可以反映肿瘤或转移性病变的基因组谱,而甲基化状态等表观遗传特征可以反映肿瘤负荷和亚型。
然后,研究团队将分析扩展到包括所有四个队列,由于与健康对照相比,肿瘤衍生的cfDNA长度更短,因此分析了四个队列中的cfDNA片段大小。
发现cfMeDIP-seq可以定性地捕捉片段长度差异,正如预期的那样,估计的片段大小分布与总生存期和无进展生存期显着相关。
与局部样本相比,在转移性前列腺癌样本中观察到显着更短的片段大小,与片段大小显着负相关。并且,与局部患者血液相比,转移灶中的ctDNA百分比更高。
接下来,揭示了转移样本中普遍存在的高甲基化。
研究团队通过cfMeDIP揭示了转移样本中广泛的高甲基化和优先重复低甲基化,检测到这种差异甲基化区域(DMR)比正常的多7.6倍。
并且,肿瘤抑制基因启动子的全局甲基化在转移性前列腺癌中显示出显着更高的甲基化。
虽然高差异甲基化区域在启动子等调节区域中富集,但低DMR则不是,经过进一步调查,发现低DMR在重复区域中特别富集。
研究团队还发现转移样本中着丝粒周围区域优先低甲基化,良性组织和原发性肿瘤样本明显的减少,着丝粒周围区域的甲基化逐渐丧失。
另外,研究团队发现显示最高倍数变化的异常 DMR是位于编码糖皮质激素受体(GR)的基因NR3C1的启动子区域。
NR3C1的启动子区域的甲基化水平与ctDNA百分比呈临界正相关,该位点较高的甲基化水平与较差的结果相关。
最后,验证DNA甲基化组可以以高精度区分转移样本和局部样本。
研究团队创建一个预测器,通过甲基化谱来区分局部和转移性肿瘤,采用102个样本进行随机测试,将50次重复的结果汇集起来进行可视化。
测试数据集上基于甲基化谱的预测器的预测准确度和接收器操作特征下的面积 (AUROC),结果显示该方法在测试数据集上具有非常高的中值精度98.9%,同时也表明了表明使用cfMeDIP-seq数据区分早期疾病和健康对照的潜力。
研究团队再次声明:与局部前列腺癌相比,转移样本普遍存在的高甲基化,并且高甲基化的优势可能是疾病特异性的,在转移性队列中观察到更高的标准偏差。
而通过ctDNA的量化可以提供有关肿瘤负荷、转移和治疗反应的信息,为未来前列腺癌的预测、治疗提供新的方法。


全国共有49家实验室报名参加,其中45家提交有效结果,仅23家通过评价活动,通过率为51.1%。

由中国临床肿瘤学会(CSCO)主办的"2020年中国临床肿瘤学年度进展研讨会"近日于线上顺利召开,会上专家学者们对2020CSCO消化道肿瘤指南更新做了总结,近几年颇受关注的免疫治疗在新版指南中地位突显,地位相较于旧版指南有所提升。
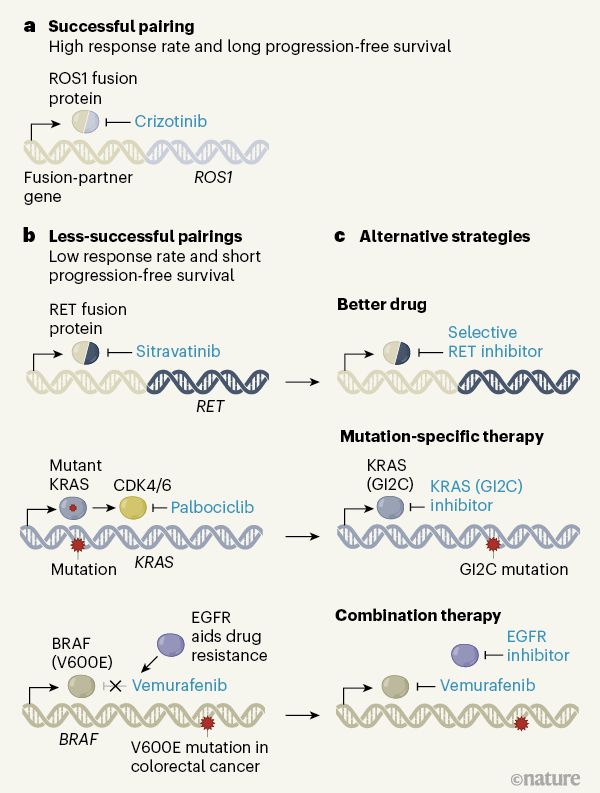
由伯明翰大学癌症研究中心英国临床试验小组领导的一项开拓性的肺癌研究强调了下一波精准医学研究,特别是治疗基因组复杂癌症需要考虑的重要因素。